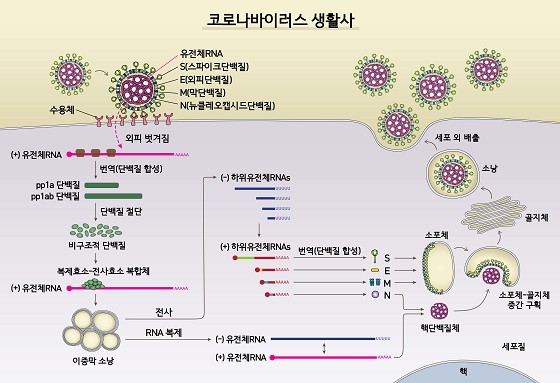
 사스코로나바이러스-2(SARS-CoV-2)의 생활사사스코로나바이러스-2의 스파이크단백질이 숙주세포의 수용체에 결합하면 바이러스가 세포 내로 진입하며, 이후 외피가 벗겨져 유전체RNA가 세포질 내에 존재하게 된다. 유전체RNA의 ORF1a/ORF1b는 pp1a와 pp1ab 단백질으로 번역되며, 이들은 단백질절단효소에 의해 절단되어 총 16개의 비구조적 단백질을 만든다. 일부 비구조적 단백질은 복제효소-전사효소 복합체(RNA 중합효소, RdRp)를 형성하여 (+)가닥에 해당하는 유전체RNA가 복제 및 전사되도록 한다. 복제 과정을 거쳐 나온 (+)가닥 유전체RNA는 추후 증식된 바이러스의 유전체가 된다. 전사 과정을 거쳐 나온 하위유전체RNA는 각각 바이러스 입자 구조를 이루는 단백질(S:스파이크단백질, E:피막단백질, M:막단백질, N:뉴클레오캡시드단백질)로 번역된다. 스파이크단백질, 피막단백질, 막단백질은 소포체 막에 들어가고, 뉴클레오캡시드단백질은 (+)유전체RNA와 합쳐져 핵단백질체가 된다. 이들은 소포체-골지체 중간 구획에서 바이러스 형태로 합쳐지고, 골지체와 소낭을 거쳐 세포 외로 배출된다/자료=IBS
사스코로나바이러스-2(SARS-CoV-2)의 생활사사스코로나바이러스-2의 스파이크단백질이 숙주세포의 수용체에 결합하면 바이러스가 세포 내로 진입하며, 이후 외피가 벗겨져 유전체RNA가 세포질 내에 존재하게 된다. 유전체RNA의 ORF1a/ORF1b는 pp1a와 pp1ab 단백질으로 번역되며, 이들은 단백질절단효소에 의해 절단되어 총 16개의 비구조적 단백질을 만든다. 일부 비구조적 단백질은 복제효소-전사효소 복합체(RNA 중합효소, RdRp)를 형성하여 (+)가닥에 해당하는 유전체RNA가 복제 및 전사되도록 한다. 복제 과정을 거쳐 나온 (+)가닥 유전체RNA는 추후 증식된 바이러스의 유전체가 된다. 전사 과정을 거쳐 나온 하위유전체RNA는 각각 바이러스 입자 구조를 이루는 단백질(S:스파이크단백질, E:피막단백질, M:막단백질, N:뉴클레오캡시드단백질)로 번역된다. 스파이크단백질, 피막단백질, 막단백질은 소포체 막에 들어가고, 뉴클레오캡시드단백질은 (+)유전체RNA와 합쳐져 핵단백질체가 된다. 이들은 소포체-골지체 중간 구획에서 바이러스 형태로 합쳐지고, 골지체와 소낭을 거쳐 세포 외로 배출된다/자료=IBS 김빛내리 IBS RNA 연구단장/사진=IBS
김빛내리 IBS RNA 연구단장/사진=IBS연구팀은 이번 연구를 통해 기존 진단 분석법으로는 확인되지 않았던 RNA들을 모두 찾았고, 바이러스 RNA에 화학적 변형이 일어나고 있다는 사실도 발견했다. 향후 바이러스 진단 및 새로운 코로나10 치료 전략을 세우는 데 필요한 단서다. 이를테면 바이러스 내부 유전자 1번과 10번 사이에 8번이 변이를 일으킨 유전자로 확인되면 이를 타깃으로 한 진단시약과 치료약물을 개발하면 된다.
김 단장은 새로 발견한 RNA들이 바이러스 복제와 숙주의 면역 반응을 조절하는 지를 알아보는 후속 연구를 이어갈 예정이다. 김 단장은 “RNA 변형은 바이러스 생존과 면역 반응과 관련이 있을 것”이라며 “이번에 발견한 RNA들과 RNA 변형은 바이러스 치료제를 개발할 때 표적으로 삼을만한 후보군이 될 것”이라고 말했다. 이어 “이번 연구를 통해 바이러스 RNA들의 각각의 위치와 양을 정확하게 파악했다”면서 “이는 진단용 유전자증폭기술(PCR)을 개선하는 데에도 도움이 될 것”이라고 덧붙였다.
한편, 연구팀은 바이러스 RNA 분석의 신뢰도를 높이기 위해 ‘나노포어 직접RNA 시퀀싱’, ‘나노볼 DNA 시퀀싱’ 등 두 가지 염기분석법을 함께 썼다. RNA 염기서열 분석은 DNA로 변환하는 과정을 거친 후 분석하는 것이 일반적이지만, ‘나노포어 직접RNA 시퀀싱’은 RNA 염기를 있는 그대로 읽을 수 있다. 즉, 원어를 번역하지 않고 직독직해하는 것과 같다. ‘나노볼 DNA 염기분석법’은 염기서열을 대용량으로 분석할 수 있다는 장점이 있다. 이를 통해 적은 용량을 분석하는 나노포어 직접RNA 시퀀싱의 약점을 보완했다.
